《科學》:媲美AlphaFold2的蛋白質結構預測新工具問世,一臺游戲計算機十分鐘出結果,完全免費
以下文章來源于學術頭條 ,作者學術頭條
如今,人工智能已經滲透到人們生活的方方面面,各種深度學習算法也越來越多地應用于各個領域。尤其在生物和醫學領域,人工智能技術可以說大放異彩,極大加速了有關生物、病理等科學的發展,而其中一項重要的應用,就是預測蛋白質結構。
蛋白質作為構成人體組織器官的支架和主要物質,在人體生命活動中起著重要作用。2020 年,DeepMind 在第 14 屆 “蛋白質結構預測關鍵評估”(CASP14)大賽中展示了轟動一時的相關成果 ——AlphaFold2,當時,該技術預測蛋白質結構的準確度排名第一。
現在,西雅圖華盛頓大學醫學院蛋白質設計研究所的研究人員,研發出一款新的深度學習工具 RoseTTAFold,不僅擁有媲美 AlphaFold2 的蛋白質結構預測超高準確度,而且更快、所需計算機處理能力更低,更重要的是,RoseTTAFold 完全免費!
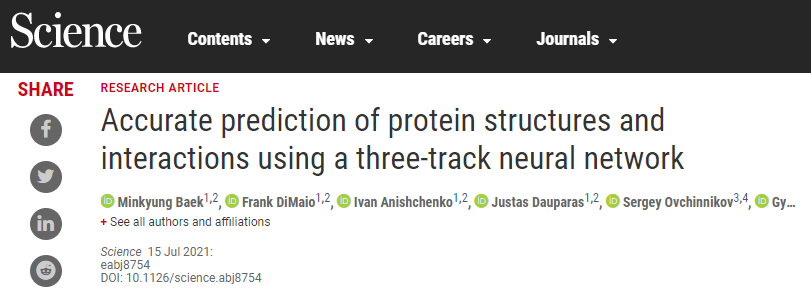
相關論文發表在最新一期的 Science 雜志上。
AlphaFold2:聲名大噪的 “前浪”
蛋白質是一切生命的物質基礎,由無數氨基酸鏈組成,它們按照特定方式折疊結合成復雜的微觀形狀,這些獨特的結構反過來又引發了生物體內幾乎所有的化學過程。因此,通過更好地了解蛋白質內部結構,科學家們可以加快開發針對癌癥、COVID-19 和數千種其它健康疾病的新療法。
圖 | 蛋白質 3D 結構(來源:Nat Commun)
Science 雜志曾指出,蛋白質折疊問題是人類在 21 世紀需要解決的 125 個科學前沿問題之一。通過蛋白質結構預測破譯 “第二遺傳密碼”,是生物學中心法則尚未揭示的奧妙之一,也是目前結構生物學面臨的一項具有挑戰性的重大基礎性研究課題。
然而,確定蛋白質的 3D 結構一直是一個難題。在過去的幾十年中,人類已經能夠利用冷凍電子顯微鏡、核磁共振或 X 射線晶體學等實驗技術確定蛋白質的基本結構,但這些技術基于大量試錯,往往需要花費數年時間,成本也非常高。
近年來,隨著人工智能技術的不斷發展,有關預測蛋白質結構的 AI 工具也越來越成熟。其中,性能最強、準確度最高的就是去年 DeepMind 在 CASP14 蛋白質結構預測評估會議上展示的 AlphaFold2。在去年的比賽中,AlphaFold2 預測的大部分結構達到了空前的準確度,不僅與實驗方法得出的結果不相上下,還遠超解析新蛋白質結構的其他方法。
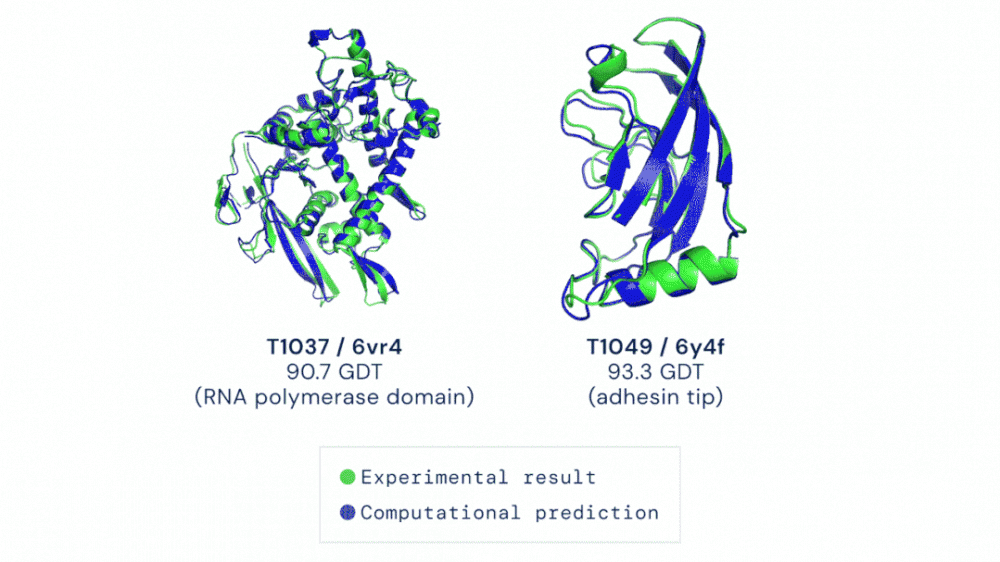
圖 | AlphaFold2 預測的蛋白結構與實驗結果幾乎一致。(來源:DeepMind)
AlphaFold2 的高性能令世人矚目,然而人們也開始思考一個問題:除了 DeepMind 這種在世界領先的深度學習公司以外,其他機構或科研團隊開發的系統中是否也可以實現這種準確性呢?
RoseTTAFold:公開免費的 “后浪”
現在,肯定的答案浮出水面。
在此次的新研究中,華盛頓大學醫學院生物化學系教授、蛋白質設計研究所所長 David Baker 領導一支計算生物學家團隊,成功開發一款名為 RoseTTAFold 的工具,基于深度學習,能夠根據有限的信息快速準確地預測出目標蛋白質的結構,達到與 AlphaFold2 不相上下的準確度。

圖 | David Baker(來源:UW)
不僅如此,RoseTTAFold 所需的計算耗能與計算時間均比 AlphaFold2 還要低:僅用一臺游戲計算機,在短短十分鐘內就可以可靠地計算出蛋白質結構。更值得注意的是,RoseTTAFold 的代碼和服務器完全免費提供給科學界!自 7 月以來,相關程序已被 140 多個獨立科研團隊從 GitHub 免費下載,來自世界各地的科學家現在正在使用 RoseTTAFold 來構建蛋白質模型,以加速相關領域的研究。
因此可以說,RoseTTAFold 不僅僅是 “免費版” 的 AlphaFold2,更是該技術領域推翻 “前浪” 的那一股 “后浪”。
整個研究學界都將受益
實際上,RoseTTAFold 是一個 “三軌” 神經網絡("three-track" neural network),這意味著它同時考慮一維蛋白質中的氨基酸序列、二維蛋白質的氨基酸如何相互作用以及蛋白質可能的三維結構。在這種架構中,一維、二維和三維信息來回流動,從而使神經網絡能夠共同推理出蛋白質的化學部分與其折疊結構之間的關系。
目前,該團隊已經使用 RoseTTAFold 計算了數百種新的蛋白質結構,其中包括許多來自人類基因組的知之甚少的蛋白質。研究人員還生成了與人類健康直接相關的蛋白質結構,包括與非正常脂質代謝、炎癥障礙和癌細胞生長相關的蛋白質結構。這些成果都表明,RoseTTAFold 可以僅用從前所需時間的很小一部分,構建出復雜生物組件的模型。
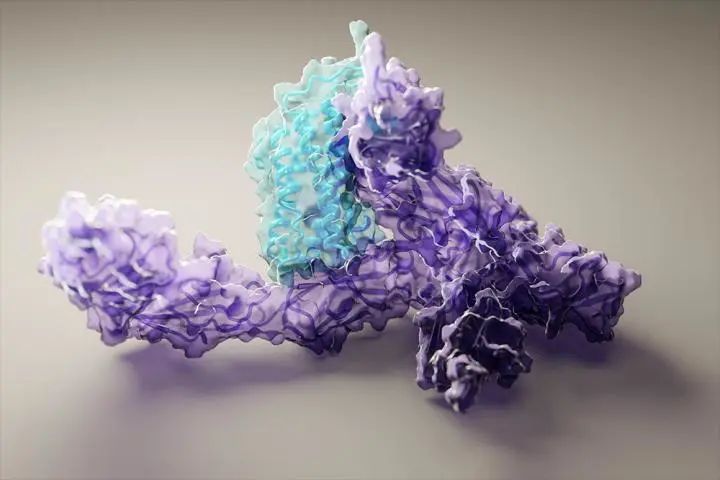
圖 | 研究人員使用 RoseTTAFold 生成了數百種新的蛋白質結構,包括人類白細胞介素 12 與其受體結合的 3D 視圖(來源:UW Medicine Institute for Protein Design)
當然,看似 “小小” 的一步,背后支撐的是整個研究團隊的努力。作為團隊主要負責人,Baker 說:“在蛋白質設計研究所這忙碌的一年中,我們設計 COVID-19 療法和疫苗并將其投入臨床試驗,同時開發出用于高精度蛋白質結構預測的 RoseTTAFold 工具。我很高興科學界已經在使用 RoseTTAFold 服務器來解決突出的生物學問題。”
可以想見的是,如此一款便捷免費、高準確度、低成本的工具必然會受到世界各地的科研人員的歡迎。團隊成員之一、博士后學者 Minkyung Baek 也表示:“我們希望 RoseTTAFold 新工具在今后,繼續使整個研究學界受益。”
參考資料:
https://science.sciencemag.org/content/early/2021/07/14/science.abj8754
https://science.sciencemag.org/content/373/6552/262
https://newsroom.uw.edu/news/accurate-protein-structure-prediction-now-accessible-all
*博客內容為網友個人發布,僅代表博主個人觀點,如有侵權請聯系工作人員刪除。