清華學者主導干濕結合“下一代細胞工廠”開源使能平臺問世!數據驅動全基因組基因型-工業表型關聯技術,賦能合成生物學高效底盤細胞設計
1976 年 1 月的一天,一位年輕的風險投資人和一位微生物學教授走進了加州大學舊金山分校(UCSF)附近的酒吧,原定十分鐘的會面時間延長到了三個小時。從那一刻起,一家改變生物技術史的公司就此誕生。他們使用 GENetic ENgineering TECHnology 的縮寫命名了這家公司——Gen-en-tech(基因泰克)。

圖丨Genentech 的聯合創始人赫伯特·博耶(Herbert Boyer)博士(左)和風險投資人羅伯特·斯萬森(Robert A.Swanson)先生(右)(來源:資料圖)
Genentech 公司首次成功地將人胰島素的 DNA 重組到大腸桿菌細胞內的質粒上,讓大腸桿菌作為細胞工廠生產出重組人胰島素,并將其產品化。從此,正式拉開了基因工程的序幕。
然而,四十多年過去了,人們在技術層面上重組表達胰島素,幾乎只做了一件事——把外源的 DNA 放進細胞,讓這個基因轉錄成 RNA,翻譯成蛋白質,然后再圍繞著該外源 DNA 的表達翻譯效率做些工程化改造。
蛋白要在宿主細胞中高效表達,其實不僅是信息傳遞。原料供應、肽鏈延長、翻譯后修飾,折疊、分泌乃至應急修復等,諸多環節都有可能影響到蛋白表達的效率。
“要讓細胞這臺精巧的蛋白質‘3D 打印機’高效率運轉,在全基因組層面有大量的基因發揮著不可或缺作用。然而,盡管我們在基礎研究的層面認識到這件事,但是,到工程層面上,還很難做到全基因組層面的工程化以提升細胞的蛋白表達效率,我們目前的認識還很淺。”清華大學張翀教授表示。
張翀是清華大學長聘副教授,國家級青年人才計劃獲得者,主要研究方向為微生物智能制造,開展高通量基因型-表型關聯原創技術與裝備的研究,包括微生物工業表型高通量表征與連續進化,全基因組規模基因及位點功能挖掘,基因型與工業表型關聯研究裝備等。

圖丨張翀(來源:張翀)
迄今為止,細胞工廠已能夠生產抗生素、氨基酸、重組蛋白、生物能源、生物塑料乃至“人造肉”,被廣泛地應用在生物制造、制****、食品、能源和農業等領域。
但是,與重組胰島素合成的案例一樣,目前人們對外源途徑改造較多,但對全基因組層面底盤細胞本身了解較少,進而制約了對其系統化工程改造的能力,細胞底盤自身的潛能還沒有被系統地挖掘。
如果把外源途徑的基因序列比作圖紙,把細胞比作車間,那么,現有的努力大多是在“圖紙”上下功夫,但是仍然十分缺乏對“車間”全局的系統認知和工程化改造的能力。
從隨機誘變到全基因組定制,多項技術催生底盤細胞“發現新大陸”

合成生物學細胞工廠構建的核心是如何通過設計合適的基因型,從而得到人們想要的工業表型。張翀教授認為,在基因組時代,科學家可通過各類公開生物學數據庫得到大量的基因型相關的測試數據,但是,真正有價值的是能得到與工業表型關聯的基因型數據。
圖丨合成生物學常關注的工業表型(來源:該團隊)
隨著分子生物學和基因工程研究方法的不斷發展,細胞工廠的構建策略經歷了不同的歷史階段。相較于早期主要通過非理性誘變育種技術獲得目標產物高產菌株的方式,20 世紀 90 年代以來,隨著分子生物學、基因工程技術的逐步引入,代謝工程學科正式創立。
代謝工程利用重組 DNA 技術對生物體中已知的代謝途徑進行有目的的設計,并對細胞內的基因網絡進行調控和優化,構建具有特定功能的細胞工廠,例如提高目的產物的產率。
然而,代謝工程指導的設計方法大多都基于已知的生物學知識,由于微生物代謝網絡中存在諸多可能對目標產物工業表型產生影響的未知因素,或稱為“生命暗物質”。這一手段獲取新知識的效率不高,細胞工廠改造過程仍然需要耗費大量的時間和精力。
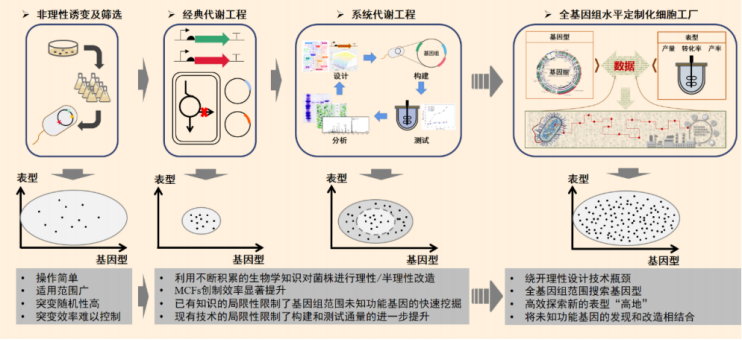
圖丨細胞工廠設計和構建發展歷程與展望 (來源:該團隊)
那么,為了讓讓細胞工廠的設計更高效,如何去解析這些“生命暗物質”呢?
“通過自有的技術平臺從全基因組層次并行研究微生物特定工業表型與基因型的關系,從而獲得大規模的基因型-工業表型關聯(genotype phenotype associations,GPA)數據集。”張翀表示。
“近年來,高通量 DNA 合成成本的降低、基因編輯技術和二代測序技術的飛躍、高通量檢測技術的成熟等多項技術的發展讓大規模的 GPA 數據集的挖掘成為可能。這些新挖掘的數據將成為基因組層面的‘新大陸’。”他說。
“新大陸”指的是新發現的跟目的工業表型相關聯的基因位點,例如他們發現很多意外的、和蛋白質合成有關系的位點,比如氧化應激(Oxidative Stress)對蛋白質合成有積極的幫助等。
“基于全基因組規模關聯圖譜獲取新知識并驗證其工業價值后,再在底盤上進行系統工程改造,將為系統提升細胞工廠效率提供一條‘發現-工程’相結合的全新路徑。”張翀說。
通過超高通量、快速、低成本的技術“三部曲”、實現全基因組規模基因型-工業表型關聯位點挖掘

張翀團隊已經成熟打造了一套技術平臺,通過該平臺,可進行超高通量、快速、低成本地對全基因組規模進行 GPA 數據集的挖掘。
該平臺的背后有三個核心技術的支撐,分別為:CRISPR 全基因組編輯技術、超高通量液滴微流控單克隆培養篩選一體化技術和合成生物傳感技術。
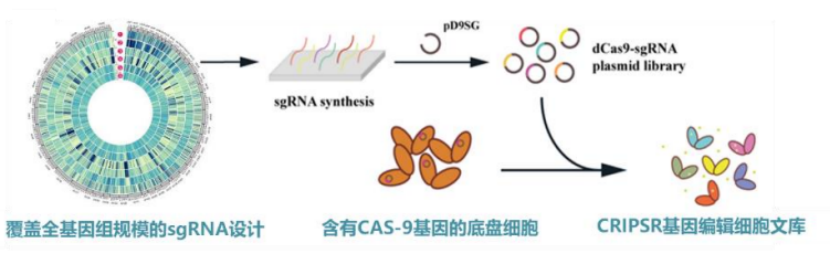
圖丨全基因組規模 CRISPR 基因干擾文庫(來源:該團隊)
第一,CRISPR 全基因組編輯技術。該團隊針對典型的工業宿主建立了細胞全基因組規模可達百萬量級 CRISPR 基因干擾文庫。這里的“干擾”,指的是把底盤細胞的基因敲低或激活,甚至進行基因編輯[1,2,3]。
張翀表示,在該平臺通過 CRISPR 編輯技術實現了“高版本”的基因型突變。這里的“高版本”是因其具備可定制、可追蹤兩大特點。也就是說,科學家可在任意位點設計 sgRNA 的干擾或編輯,并且,在表型變化后,不用測全基因組即可追蹤 sgRNA 的具體位置。
據悉,目前該團隊已有多種成熟工業底盤細胞全基因組編輯細胞文庫,并獨立開發了全基因組 sgRNA 文庫設計軟件與 web 應用程序。
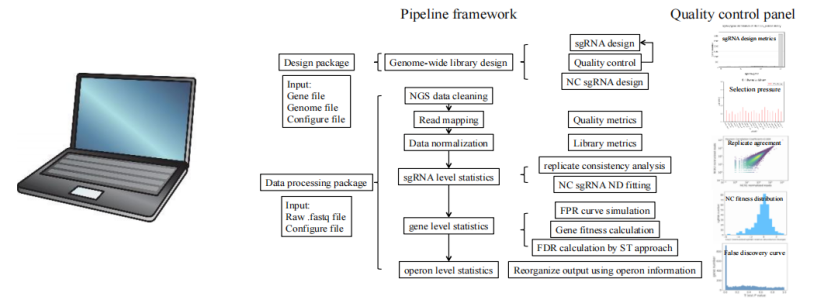
圖丨全基因組 sRNA 文庫設計軟件工具開發流程(來源:該團隊)
第二,自主研發具有百萬通量級篩選能力的液滴微流控細胞培養及篩選一體化技術。張翀團隊結合微流控技術和光電傳感與控制及自動化技術開發的“微生物微液滴培養技術 ”,可實現皮升、納升、微升級多種體積規模下的微生物液滴平行培養、生長曲線測定和適應性進化。
該平臺采用集成式單克隆培養,單次單克隆數量可超 106 個,與傳統方法相比,培養成本降低約 1000 倍 。并且,該平臺可自動換液,細胞生長狀態高度均一 ,適宜多種成熟工業微生物生長[4]。
張翀指出,通過環境條件的控制,液滴內微生物可能實現“工業相似性”培養。“這相當于實現了文庫獨立的基因型轉變為獨立的反應器,讓它生長出可獲取的目標表型。”
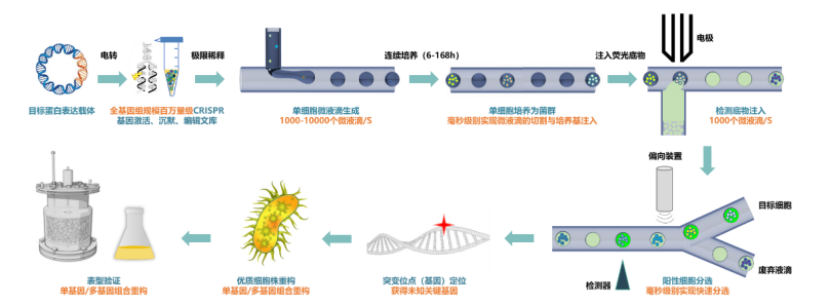
圖丨超高通量液滴微流控單克隆培養篩選一體化平臺(來源:該團隊)
第三,自主研發的高靈敏、毫秒級響應合成生物傳感器。通常,對百萬個皮升級液滴的表型進行測試,會采用定向的光學技術,尤其是熒光技術。為此,該團隊建立了一系列針對蛋白質、小分子濃度定量測試的合成生物熒光傳感技術[5,6,7]。
通過該技術,可將目標分子濃度定量地轉化成熒光信號,其靈敏度高、響應速度快,與百萬量級通量的液滴微流控系統完全兼容,為目的代謝物表型-基因型關聯圖譜的繪制奠定基礎。
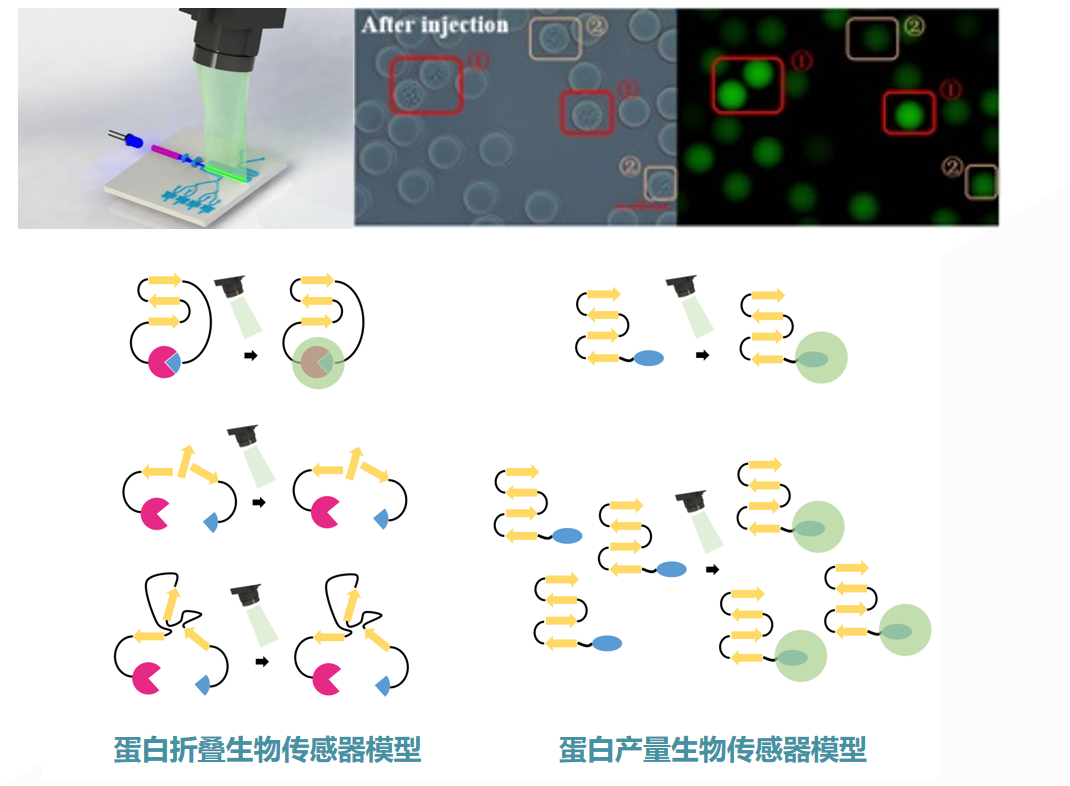
圖丨蛋白產量與折疊生物傳感器模型(來源:該團隊)
干濕結合的中心化實驗室,整合數據孤島,打造國內首個合成生物學數據開源使能平臺

目前,生物科學領域所使用的數據庫,例如 NCBI、KEGG、PDB 等,都是靠科學家團隊供應離散式數據集成形成的,其主要還是科學屬性的數據庫。“設想如果我們手頭擁有海量工業需求驅動的 GPA 數據庫,這樣就掌握著設計高效細胞工廠的核心原始數據。”張翀說。
他指出,從行業內部來看,合成生物學雖然潛力巨大,但是技術發展得很早期,還有很多問題需要解決。合成生物學是個兼具科學屬性與工程屬性的交叉學科,但是現階段,科學屬性仍然較重,工程化較弱。尤其是學術界,很多研發的方式還停留在“手工作坊”階段。
張翀認為,通過自動化和高通量技術讓科學家做實驗的過程變成中心平臺化的形式,以此來促進合成生物學從科學屬性向工程屬性轉移。一旦把這個鏈條打通,未來合成生物學就變成了純粹的信息科學和數據科學的問題。
從生物制造領域來看,細胞工廠設計是未來的發展趨勢,從現在離散式的個人實驗室變成中心化實驗室平臺,從分布式的數據向集成的大規模數據生產,這種高度標準化、高質量的數據為最后演變為AI驅動的設計提供了極大的可能性。
如果能夠利用大規模 GPA 數據集,基于數據科學手段從全基因組范圍深度挖掘傳統分子生物學手段無法發現的未知關聯基因及其位點,將有可能從數據學習的角度繞開理性設計的知識瓶頸,為提高細胞工廠設計和創建效率提供全新的研究范式。
此外,由于大規模 GPA 數據集搜索范圍更寬(全基因組),不依賴于現有知識,將有可能探索之前理性/半理性所無法達到的表型“高地”,獲得生產效率更為高效、生產性能更加優越的下一代定制化細胞工廠。
對于該平臺,張翀團隊對其規劃不僅限于對科學家的單點服務,還計劃逐漸實行學術端開源,助力多維度合成生物學的數據標準化。以高通量基因型-工業表型關聯圖譜數據驅動 AI 解析細胞工廠,打造合成生物數據使能平臺。
該平臺用工程手段推進科學研究升級,將輔助科學家實現技術轉化,并銜接行業上下游。當平臺發展到一定程度,便會積累較高的產業化勢能,將來會有多種產業化的可能性。
“科學家通過提需求,確認想要做的菌株以及表型后,我們來幫他做實驗,再將結果反饋給科學家。同時,我們希望把數據沉淀到平臺,同時開放地讓學術貢獻共同獲取,并驗證它的應用價值。未來,這會成為若干工業表型圖設計的核心驅動力。”張翀說。
據悉,該團隊已與國內重點高校相關實驗室建立廣泛合作,如清華大學、天津大學、華東理工大學、上海交通大學、江南大學、中科院上海植生所等。同時,張翀正在為該平臺進行技術授權及產業化落地,目前已在籌備成立相關公司并組建技術研發團隊,張翀教授出任首席科學家。
對該平臺的未來發展,張翀充滿期待,他稱:“我相信擁有數據的核心資源,也就掌握了細胞工廠設計的核心信息,我們希望這類關鍵數據庫生根在中國,并服務于中國本土的科研與產業。”
-End-

*博客內容為網友個人發布,僅代表博主個人觀點,如有侵權請聯系工作人員刪除。
linux操作系統文章專題:linux操作系統詳解(linux不再難懂)

